Tutoriel d'utilisation de Vesta
Le Logiciel Vesta est téléchargeable en cliquand sur le logo ci-contre : 
Créer une maille cristalline |
|||||||||||||||||
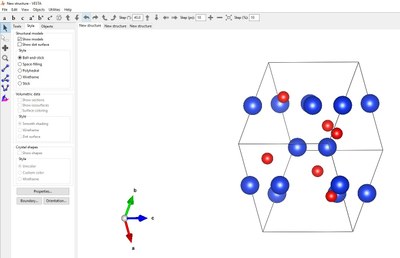
Dans ce tuto nous allons construire un quartz mais les consignes sont les mêmes pour tout type de minéral en partant de la base de données disponible plus loin.
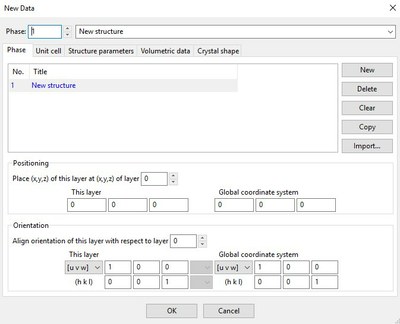
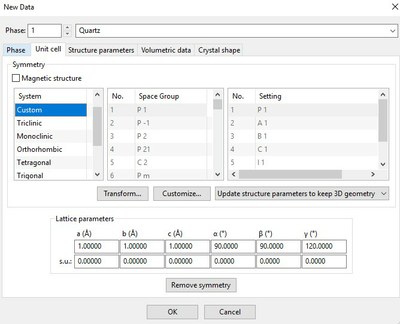
L'onglet "Unit cell" permet de sélectionner le système cristallin dans l'encart "Symmetry" puis de configurer les paramètres de réseau dans l'encart "Lattice parameter"
Pour compléter l'encart Symettry" il faut sélectionner dans les 36 colonnes les bonnes références du minéral (cf base de données)
Il n'existe qu'un seul réglage pour le quartz (colonne Setting) ce qui n'est pas le cas de la plupart des autres minéraux, il faut bien vérifier qu'il est sélectionné.
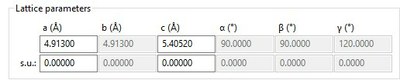
L'encart "Lattice parameters" permet d'entrer les mesures des 3 longueurs des côtés (a, b et c ; en ångström Å) et des valeurs des 3 angles (α β et γ) qui sont caractéristiques de la maille cristalline.
Dans le logiciel, seules les valeurs à compléter sont accessibles (on ne complètera que la première ligne)
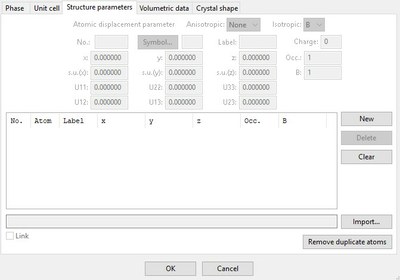
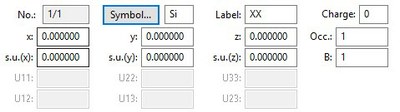
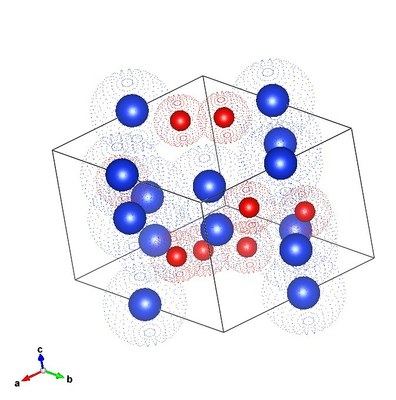
Cet onglet va vous permettre de rentrer les informations concernant la position des atomes (valeurs x, y et z). Ces données sont incluses dans la "base de données"
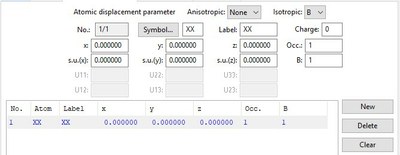
Dans le cas du quartz composé de Si et O, les positions sont les suivantes Si x=0.4699 y= 0.000 z=0.000 O x=0.4141 y= 0.2681 z=0.1188
Elle apparait en bleu, il faut la compléter pour cela :
Par défaut "New" fait apparaitre une ligne avec les paramètres du précédentsatome rentré, il ne faut pas en tenir compte et les modifier comme précédement dans les cases "Symbol", "Label" et les valeurs de "x, y et z"
|
|
||||||||||||||||
Etudier la maille |
|||||||||||||||||
|
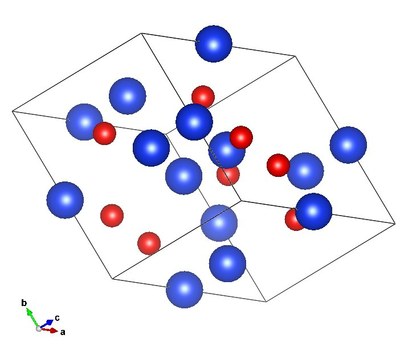
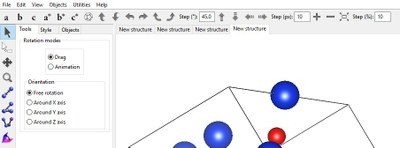
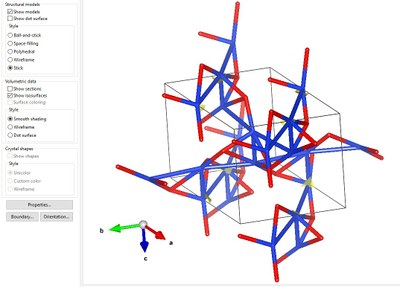
A l'aide de la souris il est possible de faire tourner la maille pour l'orienter selon les différents axes, les petites flèches abc en bas à gauche de l'écran permettent de se repérer dans les rotations.
Différents outils sont disponibles
Le menu "Tool" permet de contrôle la rotation du cristal manuellement "Drag" ou seul à l'aide du logiciel "animation" en choisissant l'axe de rotation, libre ou selon un des axes de la maille. Le menu "Style" contrôle les paramètres d'affichage.
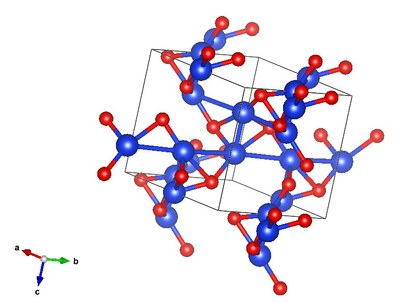
Dans le menu "Style" il est aussi possible de modifier l'apparence : "Space filing", "Ball-and-stick" ...; cependant une manip est nécéssaire afin de faire apparaitre les liaisons inter-atomiques qui n'apparaissent pas avec la méthode de construction simple proposée ici. Pour faire apparaitre les liaions il faut standardiser les données :
Cet affichage permet de comprendre les liaisons avec les atomes des mailles voisines et de comprendre que certains atomes sont partagés entre plusieurs mailles (voir plus loin)
|
|
||||||||||||||||
Réaliser des mesures |
|||||||||||||||||
|
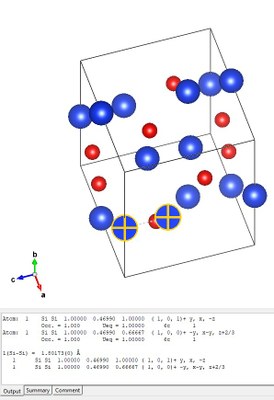
Ces mesures peuvent être réaliser avec ou sans la version standardisée. Mesurer la distance entre 2 atomes
La distance entre les deux atome apparait dans la fenêtre sous l'image dans l'onglet "Output"
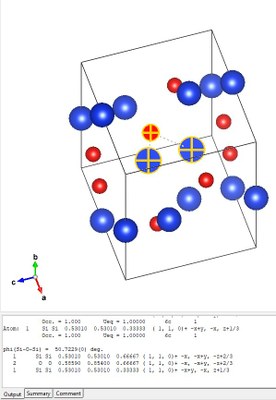
Mesurer l'angle formés entre 3 atomes
La valeur de l'angle apparait dans la fenêtre sous l'image dans l'onglet "Output" |
ici la distance mesurée entre 2 Si est de 1.80173Å
l'angle ici est de 50.7229° |
||||||||||||||||
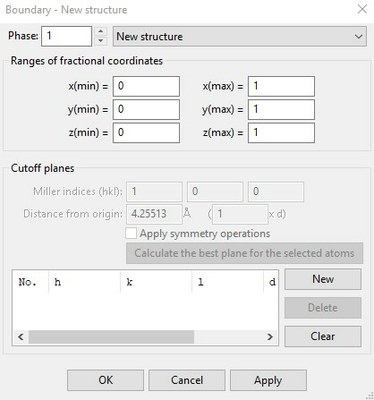
Construire un cristal en ajoutant d'autres mailles |
|||||||||||||||||
Une fenètre apparait dans laquelle il va falloir renseigner les cases de la section "Range of fractional coordinates"
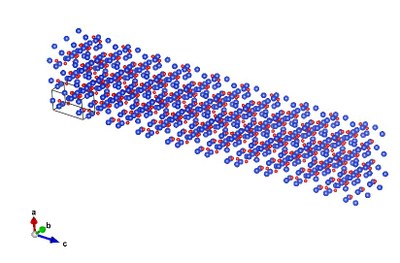
On observe ainsi les différentes mailles ajouté à la maille initiale, toujours encadrée) |
|
||||||||||||||||
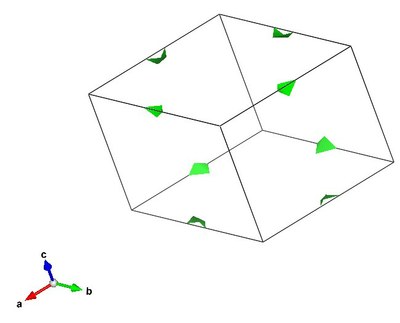
Etudier les atomes partagés entre différentes mailles |
|||||||||||||||||
Il est aussi possible de jouer dur l'opacité
|
|
||||||||||||||||
Remarques complémentaires :
- Ce logiciel permet par ailleur de visualiser de nombreux formats de molécules (cif, pmb, ...) et aussi des molécules organiques, cependant celles ci étant très volumineuses, le logiciel peut être un peu lent.
- Les molécules construites avec ce logiciel sont enregistrables et visualisables sur rastop.